β-内酰胺酶依赖和独立的进化路径导致高水平氨苄西林耐药性
作者:周李伟 时间:2024-09-13 点击数:
临床分离株中β -内酰胺耐药的发生率是一个严重的健康问题。研究抗生素耐药出现的关键方法是适应性实验室进化。然而,就β -内酰胺类抗生素氨苄西林而言,在实验室环境中进化的细菌并不能再现临床样的耐药水平,这导致了无法确定它的主要进化路径及其对遗传背景的依赖性。2024年6月10日,Rotem Gross等人在nature communications(IF=14.7)上发表了题为“Beta-lactamase dependent and independent evolutionary paths to high-level ampicillin resistance”的研究论文。
在该项研究中,他们使用微生物进化和生长竞争(MEGA-plate)板选择具有不同程度耐氨苄西林的大肠杆菌突变体。耐药菌株的全基因组测序显示,氨苄西林耐药性是通过单点突变和编码β -内酰胺酶AmpC基因扩增的组合获得的。然而,阻断AmpC介导的耐药揭示了潜在的适应途径:AmpC缺失的菌株能够通过编码外排泵、转录调节因子和孔蛋白的多药耐药基因的变化组合来适应。他们的研究结果表明,不同基因突变的组合,可以在大群体规模下获得,甚至可以独立于β -内酰胺酶驱动对氨苄西林的高水平耐药。
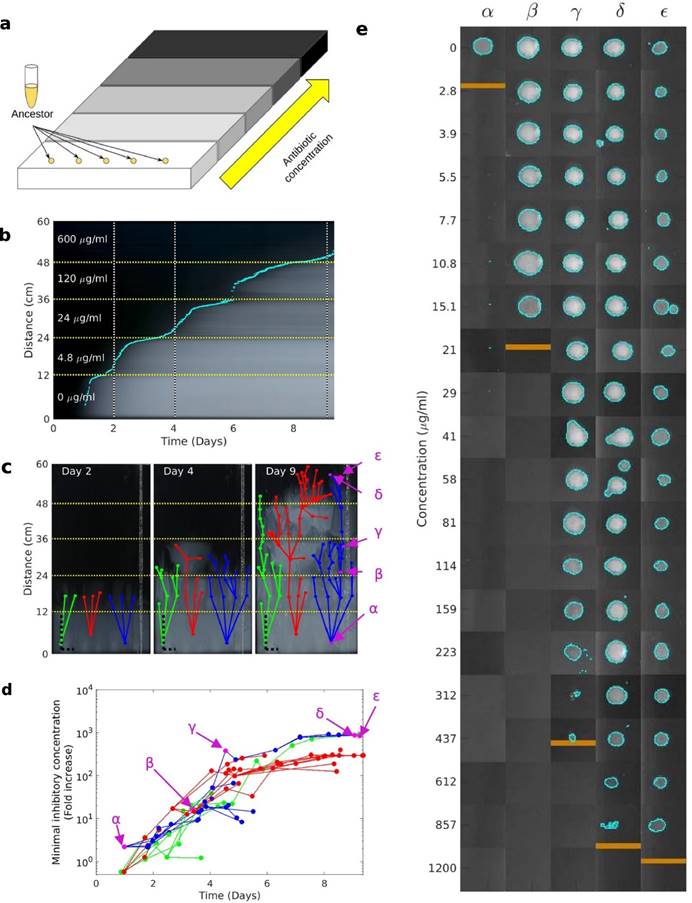
图1 MEGA 板实验设置允许高水平氨苄青霉素耐药性的演变
a MEGA 板设置的示意图,其中细菌逆着氨苄青霉素浓度增加的逐步梯度游泳。b MEGA 板实验 A 的运动图,显示每个时间点、距起点线的每个距离的像素强度的第 90 个百分位数。青色点表示细菌在每个时间点到达的最远点,由图像强度推断。垂直黑白虚线表示对应于 (c) 中呈现的静止图像的三个时间点(第 2、4 和 9 天)。c MEGA 板在三个时间点(第 2 天、第 4 天和第 9 天)的静止图像,指示从三个不同谱系(红色、绿色和蓝色连接点)采样的分离株的位置。出于演示目的,图像沿水平轴压缩,如水平和垂直黑色虚线刻度所示,均代表 10 厘米。d 在 MEGA 板上进化的三个不同谱系(绿色、红色和蓝色)的分离株的抗性水平随时间的增加。在两者 (b, c) 中,浓度步骤均用黄色虚线表示。e 图 c 和 d 中所示的 5 个分离株的 MIC 测量值(标记为 ɑ、β、γ、ε、δ,对应于补充图 3、21、47、89、90。1a);青色等值线表示在抗生素梯度上检测到的菌落,MIC 由水平橙色线表示。
原文链接:https://www.nature.com/articles/s41467-024-49621-2
DOI:10.1038/s41467-024-49621-2