噬菌体是地球上数量最丰富的生物之一,专门感染细菌。为了在噬菌体丰富的环境中生存,细菌进化出了多种防御机制。Hachiman系统作为一种新型的原核抗噬菌体防御系统,由HamA和HamB两种蛋白组成,分别含有DUF1837和解旋酶结构域。然而,其防御机制尚未完全阐明。近日,中国华中农业大学的研究团队Yongqing Cui等人在《nature communications》上发表了题为“ Bacterial Hachiman complex executes DNA cleavage for antiphage defense”的研究论文,展示了一项关于细菌Hachiman复合物的研究成果,揭示了这一新型原核抗噬菌体防御系统的分子机制。
该研究团队通过冷冻电镜(cryo-EM)解析了Hachiman防御复合物的结构,发现HamA蛋白包含一个融合的Cap4核酸酶结构域。对其进行结构分析发现,HamA和HamB以1:1的比例形成稳定的异二聚体,通过三个不同的界面相互作用,HamA的N端包含一个限制性内切酶样折叠的结构域,而HamB属于SF2解旋酶家族,其中HamA的DUF1837结构域类似于PD-(D/E)XK核酸酶,但缺乏活性位点。通过功能验证进行生物信息学分析表示,催化活性丧失的DUF1837结构域能够招募其他功能域以实现抗噬菌体防御。HamA与HamB形成异二聚体HamAB,通过ATP水解和DNA切割来执行抗噬菌体防御。
该研究团队通过结构生物学和功能验证,不仅阐明了Hachiman系统的结构基础,还突出了解旋酶和核酸酶在原核免疫中的关键作用,揭示了细菌Hachiman复合物如何通过DNA切割来防御噬菌体感染。研究结果为理解细菌如何通过进化出复杂的蛋白复合物来抵御噬菌体提供了新的视角,也为开发新型抗病毒策略提供了潜在的靶点。这一发现不仅加深了我们对细菌免疫系统的理解,还为抗病毒研究提供了新的思路。

图 1.Hachiman 系统提供对不同噬菌体的防御
a 来自大肠杆菌菌株 K-12 的 Hachiman 系统基因盒示意图。b 大肠杆菌菌株 MG1655 (ΔhamAB) 的抗噬菌体防御表型,表达具有天然启动子的 Hachiman 系统。通过连续稀释点试验测量折叠防御。每次测定进行两次重复(详细数据见补充图 1)。表达 Hachiman 系统或空载体的大肠杆菌 MG1655 菌株的生长曲线,感染噬菌体 T1 (c) 或噬菌体 TP041 (d),MOI 为 0、0.02 或 2。数据是 3 次技术重复的 SD 均值±,代表了两个独立实验。e 来自大肠杆菌 str. K-12 基因组的 Hachiman 系统的 HamA 和 HamB 蛋白的结构域织。f HamAB 复合物的冷冻电镜密度图。g HamAB 复合物结构模型的漫画。蓝色,HamA-Cap4 结构域;绿色,HamA-DUF1837;紫色,HamB-NTD; Brown,RecA-1 结构域;黄色,RecA-2;青色,HamB-C 螺旋。
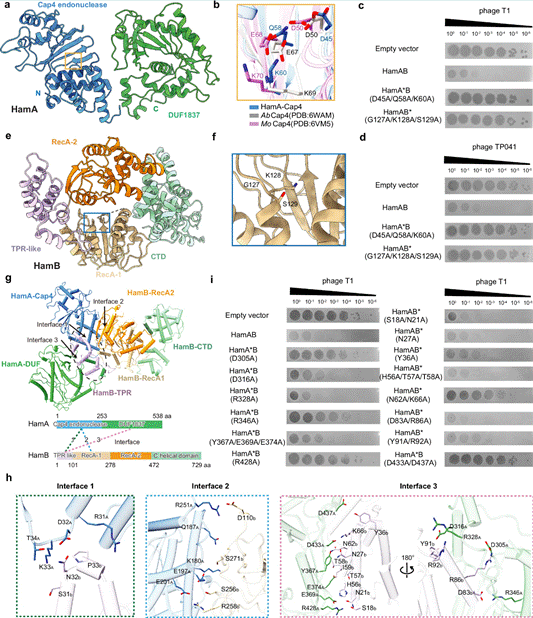
图 2.HamAB 复合物的冷冻电镜结构
a HamAB 复合物中 HamA 的卡通示意图和冷冻电镜结构。HamA 包含一个 N 端 Cap4 核酸内切酶结构域(蓝色)和一个 C 端 DUF1837 结构域(绿色)。b Cap4 核酸内切酶结构域核酸酶活性位点的放大视图。噬菌体 T1 (c) 和噬菌体 TP041 (d) 感染对含有空载体、Hachiman 系统或突变 Hachiman 系统的大肠杆菌菌株 MG1655 进行噬菌体斑点试验,具有三个生物学重复。e HamAB 复合物中 HamB 的卡通示意图和冷冻电镜结构。f 介导 HamB ATP 结合的关键残基的放大视图。g HamA 和 HamB 之间的相互作用。插图表示三个关键接口(接口 1、2 和 3)。h 界面 1、界面 2 和界面 3 的放大视图,在界面上显示关键残留物。i 表达野生型 Hachiman 系统(HamAB) 或表达突变型 HamA/HamB 的大肠杆菌 MG1655 上 T1 噬菌体感染的噬菌体斑点试验,有两个生物学重复。
原文链接:https://www.nature.com/articles/s41467-025-57851-1