许多非抗生素药物具有强大的抗菌活性,在长期用药的情况下可能会诱发多重耐药菌,从而削弱抗生素的疗效。然而这种抗菌机制在很大程度上仍然未知。2024年3月,美国马萨诸塞大学的Amir Mitchell团队在 Science(IF=56.9)发表了题为“Antibacterial activity of nonantibiotics is orthogonal to standard antibiotics”的研究论文,提出了非抗生素和抗生素的抗菌机制具有正交性,即它们通过不同的机制发挥抗菌作用。该研究团队通过对1280种药物(包括226种抗生素和1054种非抗生素)进行筛查,筛选出对大肠杆菌生长有影响的176种非抗生素和142种抗生素。随后利用集合基因筛选方法分析了药物特异性抗菌作用背后的200万种基因-药物相互作用。基于网络的药物-药物相似性分析表明,抗生素聚集成与其既定类别作用模式一致的模块,而非抗生素与抗生素之间没有联系,一些非抗生素之间存在关联,半数的非抗生素聚集在单独的模块中。这一模块化的网络结构可能揭示了存在尚未开发但作用强大的抑制细菌生长的细胞靶点,并为进一步研究提供了多种先导分子。这一发现对于理解微生物群与人类健康之间的关系、探索新的抗菌策略以及应对抗生素耐药性的全球挑战具有重要意义。
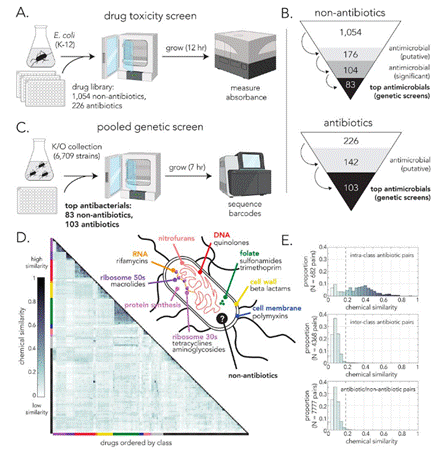
图1.筛选抗菌药物。(A)药物筛选概述 (B)非抗生素和抗生素药物的抗菌活性筛选结果 (C)汇总基因筛选概述 (D)所有药物的两两化学相似性 (E)各组药物的两两化学相似性直方图。
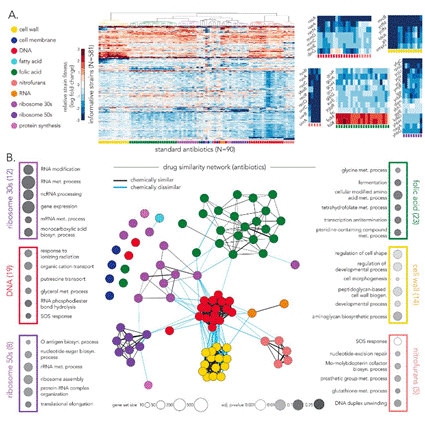
图2.基因筛选捕捉抗生素的作用机制,揭示类间的相互作用。(一) 基于信息敲除适应度评分的抗生素分层聚类 (B)信息敲除对抗生素药物相似性的网络表示。
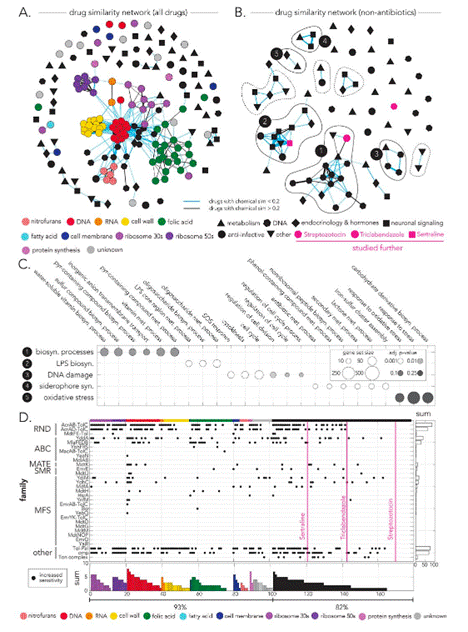
图3.非抗生素药物抗菌活性的机制。(A) 574个抗生素信息敲除对所有药物相似性的网络表示 (B)非抗生素药物相似性的网络表示 (C)氧化石墨烯生物工艺条件下非抗生素簇的功能富集。圆形大小和阴影分别表示基因集大小和统计显著性。(D)敲除对单个药物的中心药物运输和膜透性系统的影响。
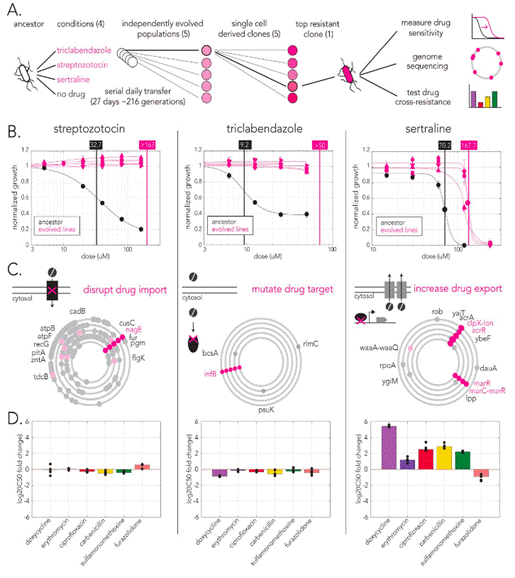
图4.对非抗生素的进化抗性暴露了新的药物靶点和附带的交叉抗性。(A)进化实验及下游分析概述 (B)耐药性在所有文化和药物中进化 (C)全基因组测序结果揭示了进化抗性的三种替代策略 (D)舍曲林进化菌株对多种抗生素耐药,而三氯苄唑和链状佐菌素进化菌株没有交叉耐药。图中显示了进化后的克隆体对五种抗生素的耐药性增加(IC50倍变化)。条形图代表所有进化克隆的中位数。点代表每个克隆的三个生物复制的平均值。条形颜色表示抗生素类别。
原文链接:https://www.science.org/doi/10.1126/science.adk7368?url_ver=Z39.88-2003&rfr_id=ori:rid:crossref.org&rfr_dat=cr_pub%20%200pubmed
DOI:10.1126/SCIENCE.ADK7368